OBJECTIVE.
東京大学生産技術研究所
立教大学
日本大学
みずほ情報総研株式会社
東京大学の加藤千幸教授、立教大学の望月祐志教授、日本大学の福澤薫助教、みずほ情報総研の加藤幸一郎コンサルタントらの研究グループは、生体親和材料であるハイドロキシアパタイトと微小タンパク質(人造ペプチド)の相互作用の系統的な大規模計算解析に世界で初めて成功しました。この成果は、東京大学生産技術研究所を拠点とする、文部科学省次世代IT 基盤構築のための研究開発「イノベーション基盤シミュレーションソフトウェアの研究開発」、ならびに文部科学省「HPCI戦略プログラム」分野4次世代ものづくりのプロジェクト(共に研究代表者は加藤千幸教授)において独自開発を進めてきたFMO計算プログラム『ABINIT-MP』とその可視化インターフェース『BioStationViewer』を用いて初めて可能になったものです。
このシミュレーション成果は、歯科治療における接着材料の開発に応用することが期待されるばかりでなく、骨形成メカニズムの解明や安全性の高い歯科・外科用インプラントの設計、ハイドロキシアパタイトを表面に有するバイオセンサーでの検出過程のシミュレーションなどのナノバイオテクノロジー分野の研究・設計に幅広く役立つと期待されます。また今回の開発によって、ABINIT-MP/BioStationViewerは、シリカの様な共有結合性固体に加えてヒドロキシアパタイトの様なイオン結合性固体の両方に対応することが可能になりましたので、ナノバイオ界面の設計ツールとしての完成度も高められています。
発表ポイント
立教大学
日本大学
みずほ情報総研株式会社
東京大学の加藤千幸教授、立教大学の望月祐志教授、日本大学の福澤薫助教、みずほ情報総研の加藤幸一郎コンサルタントらの研究グループは、生体親和材料であるハイドロキシアパタイトと微小タンパク質(人造ペプチド)の相互作用の系統的な大規模計算解析に世界で初めて成功しました。この成果は、東京大学生産技術研究所を拠点とする、文部科学省次世代IT 基盤構築のための研究開発「イノベーション基盤シミュレーションソフトウェアの研究開発」、ならびに文部科学省「HPCI戦略プログラム」分野4次世代ものづくりのプロジェクト(共に研究代表者は加藤千幸教授)において独自開発を進めてきたFMO計算プログラム『ABINIT-MP』とその可視化インターフェース『BioStationViewer』を用いて初めて可能になったものです。
このシミュレーション成果は、歯科治療における接着材料の開発に応用することが期待されるばかりでなく、骨形成メカニズムの解明や安全性の高い歯科・外科用インプラントの設計、ハイドロキシアパタイトを表面に有するバイオセンサーでの検出過程のシミュレーションなどのナノバイオテクノロジー分野の研究・設計に幅広く役立つと期待されます。また今回の開発によって、ABINIT-MP/BioStationViewerは、シリカの様な共有結合性固体に加えてヒドロキシアパタイトの様なイオン結合性固体の両方に対応することが可能になりましたので、ナノバイオ界面の設計ツールとしての完成度も高められています。
発表ポイント
(1)フラグメント分子軌道(FMO)法(注1)のための独自開発の並列化プログラムABINIT-MPと可視化解析システムBioStationViewer(注2)を用いて、1.4千原子から成る巨大なハイドロキシアパタイト結晶と接着性の微小タンパク質(ペプチド)との相互作用を電子状態レベルで詳細に解析することに成功しました。
(2)ハイドロキシアパタイト表面とペプチドとの親和性に対する電子的なメカニズムを世界で初めて明らかにしました。歯科学における接着性ペプチドの機能デザインなどに役立つと期待されます。
(3)今回のシミュレーション手法は、歯や骨の治療に関わるばかりでなく、ナノバイオテクノロジーの諸問題に展開することが出来ます。また、並列計算資源の利活用により、安全なインプラント、高感度なバイオセンサー、機能性ナノ粒子の表面に関する合理的設計に有用な実用ツールとなります。
研究の背景および技術的内容
本研究グループでは、これまで、独自に開発してきた並列化FMO計算プログラムABINIT-MPに4体までのフラグメント展開を行うFMO4法[5](注3)を実装し、4体補正によって計算精度が飛躍的に向上することを実証しました。FMO4法によればダイヤモンド、シリコン、シリカなどのバンドギャップを有する3次元の固体結晶の扱いも精度を保持して可能となることを示し[5]、大規模なシリカ結晶表面への微小タンパク質(人造ペプチド)の吸着状態の量子論的な解析に、FMO4計算によって世界で初めて成功しています[6]。
一方、固体と生体分子の相互作用の解析は、インプラントの表面改質による生体親和性の向上、ナノ粒子による薬剤投与(ドラッグデリバリ)、生体結晶析出(バイオミネラリゼーション)などの設計製造に係る医療工学や生物工学、さらには極微量でのタンパク質やDNAの検出を意図した高感度バイオセンサーといったデバイス類を開発する応用物理学の分野まで、いわゆるナノバイオテクノロジーの境界/界面の問題として、近年重要視されています。ナノバイオテクノロジーのシミュレーションでも、古典分子動力学もしくは古典/量子論混成モデリングが使われてきていますが、古典分子動力学法では電荷移動や化学反応などの電子論的な描像は捉えることが出来ず、古典/量子論混成においては量子論領域が広範囲となるために実際の計算は困難です。また、密度汎関数計算も周期性を課す場合には扱いが難しくなります。そのため、他の手法での文献[6]と同様の解析は現時点では困難であり、文献[6]はナノバイオテクノロジー分野におけるFMO計算の一里塚となりました。(前回、シリカとペプチドのFMO4計算による相互作用解析をまとめた文献[6]の研究成果を2013年4月30日にプレス発表しています。)
今回[7]は、FMO法のナノバイオテクノロジーの境界/界面への新たな適用として、生体親和性材料であるハイドロキシアパタイトの巨大結晶と人造ペプチド[8,9]との詳細な相互作用解析に世界で初めて成功しました。ハイドロキシアパタイトは歯や骨の主要な無機成分であり、表面のエナメル質と特異的に接着するペプチド分子を、計算結果を利用しつつ効率的に見出すことで、歯科学におけるより高性能な接着材料の設計や骨形成を促進する骨補填材料の開発などに広く役立つと期待されます。これは、プログラムシステムとしてのABINIT-MP、随伴する可視化インターフェースBioStationViewerのツールとしての進歩を示すものです。
具体的な計算内容
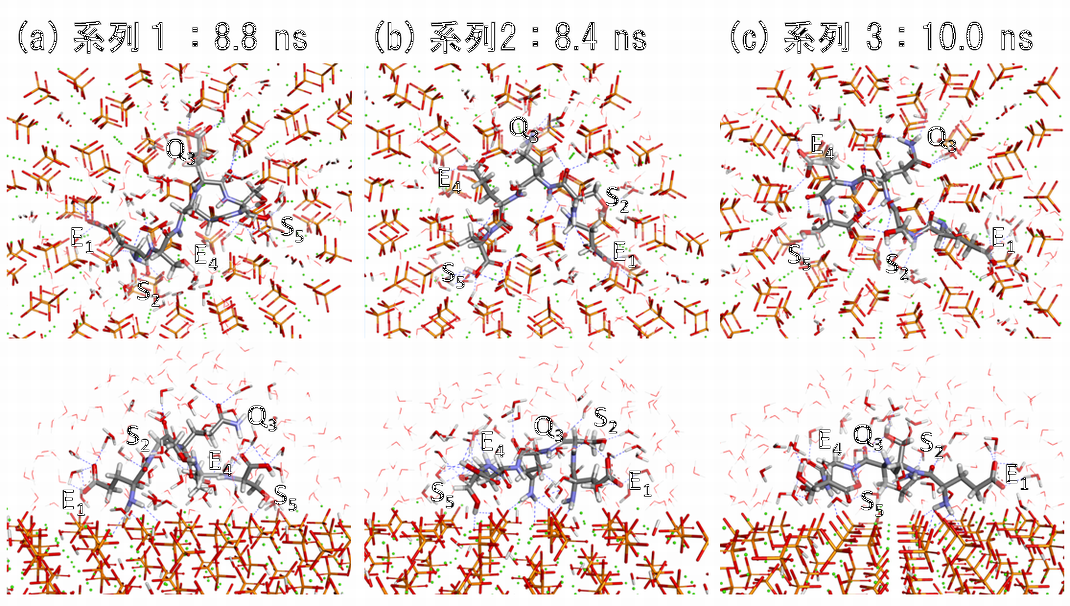
図1:アパタイト表面に吸着するペプチドの分子動力学計算でのスナップショット
図2は、各アミノ酸残基とアパタイト側の相互作用の統計的平均エネルギーをIFIE値で示したものであり、共通して末端部のセリン(S5; Ser5)が最も顕著に吸着における安定化相互作用に寄与していることが分かりました。そして、電子密度の解析により、表面で隣接するリン酸イオンからの電荷移動が安定化相互作用に本質的であることを明らかにしました。一方、構造揺らぎを考慮することで、セリン(Ser3, Ser5)やグルタミン酸(Glu1, Glu4)の様な同じアミノ酸残基であっても、構造に応じて安定化相互作用が大きく変化することも分かりました。Heら[8]は、実験的な見地からセリンとグルタミン酸とアパタイト側のイオンとの静電的相互作用による安定化を推察していましたが、今回の計算からはアミノ酸の種類だけでなく、電荷移動の効果やハイドロキシアパタイト表面側との相対的な位置関係も重要であることが新たに示されました。
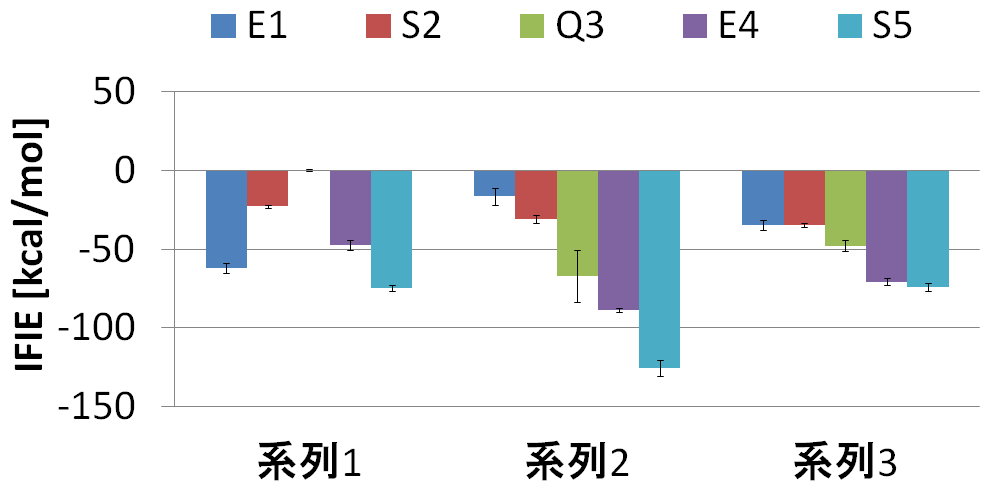
図2:5つのアミノ酸残基とアパタイトとの相互作用エネルギー
社会的意義・今後の予定
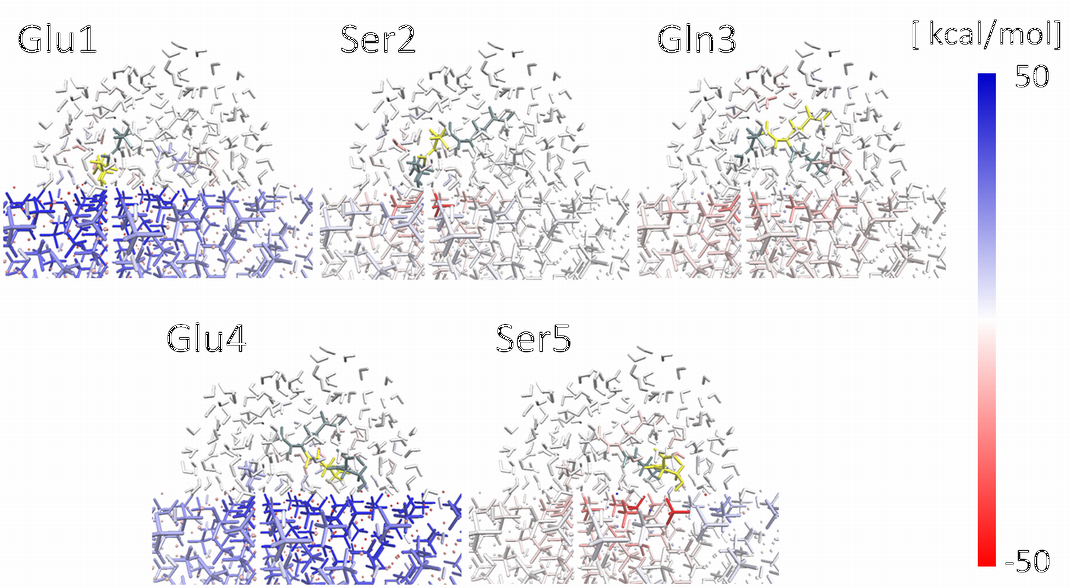
図3:系列1のアミノ酸残基毎の遮蔽補正済の相互作用エネルギーの可視化(黄色が注目するアミノ酸残基、分子動力学のスナップショット時刻は図1と同じ)
今後の応用の1つとして、歯表面と歯科材料との結合を誘導するプライマーとしてのペプチドの機能デザインなどへの展開が考えられます。また別の方向性としては、「京」に代表される高性能計算機資源(HPCI)を活かし、アルブミンやコラーゲンなどの実タンパク質がハイドロキシアパタイト表面に吸着する様子をシミュレーションすることが考えられ、骨形成メカニズムの解明や高品質なインプラント、高感度のバイオセンサー、あるいは薬品輸送用のナノ粒子の表面設計への貢献が期待されます。この際、本質的には、多数の構造サンプルを扱って統計的に処理しますが、「京」やポスト「京」の超並列計算機を使うことで高速実行が可能となります。
さらには、アミノ酸置換による「人工変異」を与えた一連のタンパク質を計算機の中で「生成」し、それらと表面との相互作用を系統的にシミュレーションし、吸着能の高い人造タンパク質を選択することも可能となります。その際、計算によって得られる膨大な数値データを解釈し、価値のある「法則」を見出す上で機械学習的なアプローチも今後は必要になると思われます。
一方で、ABINIT-MP/BioStationViewerの従来の応用ターゲットである創薬分野に関しては、H27年度「京」産業利用(実証利用)研究課題「HPCIを活用したFMO創薬プラットフォームの構築(代表者:福澤薫)」において、製薬企業10社を含むコンソーシアム形式での産業利用を推進しています。これからも、FMO計算による研究成果の産業界への展開および普及活動を、創薬やナノバイオテクノロジーなどの多分野に対して進めていく予定です。
本研究グループでは、ABINIT-MPによるFMO計算が、これまでの創薬分野だけでなく「ものづくり」の様々な文脈でも活用され得ることを今後も実証していきたいと考えています。
発表雑誌
用語解説
タンパク質のような巨大な分子系を小さなフラグメント(アミノ酸残基など)に分けて、並列処理を駆使して計算コストを低減する分割-統合系の手法[1]の中の代表的な計算法。1999年に北浦ら[2]が提案し、この15年ほどで急速に技術開発と応用計算が展開されている。FMO計算からは、フラグメント間の相互作用エネルギー(IFIEやPIEと呼ばれる)が得られ、系内部の相互作用の解析に好適である[3,4]。
注2:BioStationViewer
FMO計算では結合を切断してフラグメントに分割する際に注意が必要であり、タンパク質のように大型の系では人間が手動で行うとミスが発生しやすいために、可視的な支援ツール(GUI)の利用が望ましい。また、IFIEなどの結果データも膨大な量となるためにログファイルなどから手動で拾い、物理化学的な意味を理解することは困難であり、やはり可視化インターフェースの利用が有用である。BioStationViewerは、ABINIT-MPと共に開発が進められてきたGUIで使い勝手に大変優れ、応用計算のワークフローを強力にサポートする[4]。
注3:FMO4法
フラグメントの展開を4体(テトラマー)まで拡張した手法で、分割しないMO計算に比してもエネルギーの誤差が少ない[5]。FMO4法は、ABINIT-MP[4]にのみ実装されており、高速化の工夫により通常の2体展開に比して10倍程度のコスト増で計算出来る。FMO4計算によれば、リガンドの複数分割とアミノ酸残基の主鎖・側鎖分割を伴う高解像度の解析も可能である[4]。また、ダイヤモンド、シリコン、シリカなどの3次元の結合ネットワークを持つ固体[5,6]、さらにハイドロキシアパタイトのようなイオン結晶も扱うことが出来る[7]。
注4:計算時間
ABINIT-MPは、高い並列処理親和性と共に演算カーネルの高速化も進んでおり、「京」にも対応している(HPCI-戦略分野4のライブラリとして登録済み)。今回は、小規模なサーバを用いて1構造あたり3時間でFMO4-MP2エネルギー計算を処理している。今後、「京」のような超並列計算機を利用すれば、タンパク質とより大型の結晶モデルを使った実在系のモデルを効率よく実行出来、ナノバイオテクノロジー分野での合理的設計に役立つと期待される。
注5:SCIFIE
イオン性が強い系ではIFIEが過大評価される傾向にあるが、SCIFIEではフラグメント間の遮蔽を統計的に補正する手法によって遠方のフラグメントで期待される減衰効果を含めた値を得ることが出来る[10]。
参考文献
[2]K. Kitaura, E. Ikeo, T. Asada, T. Nakano, M. Uebayasi, Chem. Phys. Lett. 313 (1999) 701.
[3]D.G. Fedorov, T. Nagata, K. Kitaura, Phys. Chem. Chem. Phys. 14 (2012) 7562.
[4]S. Tanaka, Y. Mochizuki, Y. Komeiji, Y. Okiyama, K. Fukuzawa, Phys. Chem. Chem. Phys. 16 (2014) 10310.
[5]T. Nakano, Y. Mochizuki, K. Yamashita, C. Watanabe, K. Fukuzawa, K. Segawa, Y. Okiyama, T. Tsukamoto, S. Tanaka, Chem. Phys. Lett. 523 (2012) 128.
[6]Y. Okiyama, T. Tsukamoto, C. Watanabe, K. Fukuzawa, S. Tanaka, Y. Mochizuki, Chem. Phys. Lett. 566 (2013) 25.
[7]K. Kato, K. Fukuzawa, Y. Mochizuki, Chem. Phys. Lett., in press (DOI information: 10.1016/j.cplett.2015.03.057).
[8]G. He, T. Dahl, A. Veis, A. George, Nat. Mater. 2 (2003) 552.
[9]T. Tsuji, Y. Oaki, M. Yoshinari, T. Kato, K. Shiba, Chem. Comm. 46 (2010) 6675.
[10]S. Tanaka, C. Watanabe, Y. Okiyama, Chem. Phys. Lett. 556 (2013) 272.
研究支援
お問い合わせ
研究内容について
東京大学 生産技術研究所 教授
担当:本研究の推進(「HPCI戦略プログラム」分野4のリーダー)
〒153-8505 東京都目黒区駒場4-6-1
電話:03-5452-6190 ファックス:03-5452-6191
望月 祐志(モチヅキ ユウジ)
立教大学 理学部 化学科 教授
担当:全体の企画・取りまとめ(FMO計算法、ABINIT-MPの開発リーダー)
〒171-8501 東京都豊島区西池袋3-34-1
電話:03-3985-2407 ファックス:03-3985-2407
福澤 薫(フクザワ カオリ)
日本大学松戸歯学部 化学教室 助教
担当:本研究の解析(生体分子の応用計算、BioStationViewerのデザイン)
〒271-8587 千葉県松戸市栄町西2-870-1
電話:047-360-9316 ファックス:047-360-9316
加藤 幸一郎(カトウ コウイチロウ)
みずほ情報総研株式会社 サイエンスソリューション部 コンサルタント
担当:本研究の実施と解析(表面・固体物理系の応用計算)
〒101-8443 東京都千代田区神田錦町2-3
電話:03-5281-5271 ファックス:03-5281-5414
研究内容以外のお問い合わせ
〒153-8505 目黒区駒場4-6-1
電話:03-5452-6017 ファックス:03-5452-6071
E-mail:koho@iis.u-tokyo.ac.jp
学校法人立教学院 企画部広報課(担当:宇野)
〒171-8501 東京都豊島区西池袋3-34-1
電話:03-3985-2202 ファックス:03-3985-2827
E-mail:koho@rikkyo.ac.jp
日本大学松戸歯学部 庶務課(担当:山室)
〒271-8587千葉県松戸市栄町西2-870-1
電話:047-360-9203 ファックス:047-364-6295
E-mail:yamamuro.takashi56@nihon-u.ac.jp
みずほ情報総研株式会社 広報室(担当:石原)
〒101-8443 東京都千代田区神田錦町2-3
電話:03-5281-7548 ファックス:03-3259-2185
E-mail:info@mizuho-ir.co.jp